NAR:基于人工智能技术在非编码DNA中发现新型癌症驱动突变
2024-08-02 测序中国 测序中国 发表于上海
这项研究颠覆了人们对DNA的传统认识,将研究视角扩展到了之前被忽视的非编码区域。
近日,澳大利亚Garvan医学研究所的Amanda Khoury&Susan J Clark团队运用人工智能技术,在DNA序列中被认为无用的“垃圾”区域——非编码DNA中发现了若干潜在的癌症驱动因素,这项研究颠覆了人们对DNA的传统认识,将研究视角扩展到了之前被忽视的非编码区域。这项最新研究成果以“Machine learning enables pan-cancer identification of mutational hotspots at persistent CTCF binding sites” 为题发表在Nucleic Acids Research上。

据悉,该研究揭示了一类以前未被充分研究的基因组区域的突变现象,这些突变可能参与了至少12种不同类型癌症的发生与发展过程,包括常见前列腺癌、乳腺癌和结直肠癌等,这一重大突破预示着有望在癌症早期诊断和治疗领域发现有效的新方法和途
CCCTC结合因子(CTCF)是一种绝缘体蛋白,普遍存在于真核生物中,可与高度保守的DNA基序结合并促进三维(3D)核结构和转录的调节。CTCF结合位点(CTCF-BSs)位于在非编码DNA中,其数量庞大且具有显著异质性,经常在癌症中发生突变。
已知CTCF敲低可导致CTCF-BSs的广泛丧失,称为丢失性CTCF结合位点(L-CTCF-BSs);先前研究还确定了一小类对CTCF敲低具有抗性的CTCF-BSs,称为持久性CTCF结合位点(P-CTCF-BSs)。与L-CTCF-BSs相比,P-CTCF-BSs表现出不同的遗传和表观遗传特性,具有高度结合保守性,并可能调控细胞型的构成性3D染色质结构。但L-CTCF-BSs的突变频率,以及其能否导致目前仍不清楚。
为解决上述难题,研究团队开发了一种名为“CTCF-INSITE”的复杂人工智能(AI)工具,可基于实验定义的P-CTCF-BSs,预测CTCF在癌细胞中敲低后的结合持久性。值得注意的是,在所有12种癌症类型中,预测的P-CTCF-BSs显示出显著升高的突变负荷;对于预测会对CTCF结合和染色质环产生功能性影响的P-CTCF-BSs突变,其富集程度更加明显。使用体外结合实验,研究团队证实了预测具有破坏性的P-CTCF-BSs癌症突变确实降低了CTCF的结合。综上,该研究揭示了一类新的癌症特异性CTCF-BS DNA突变,并为其在泛癌症背景下对基因组组织的重要性提供了见解。
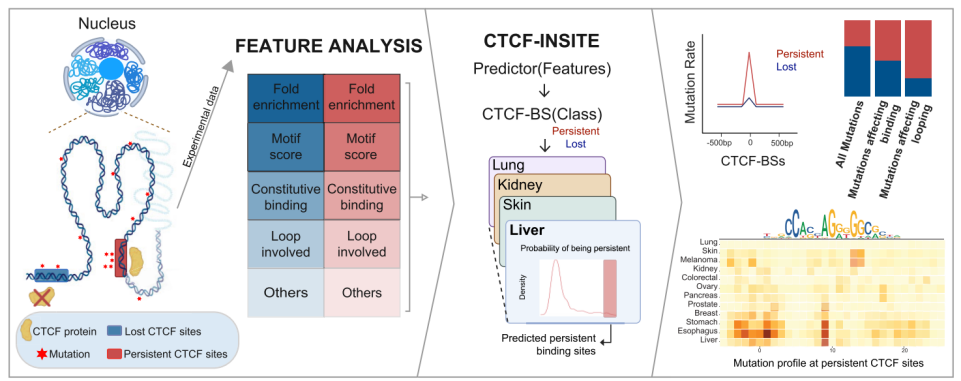
图:研究概要
P-CTCF-BSs可从不同基因组和表观基因组特征中预测
研究团队首先评估了实验定义的P-CTCF-BSs的基因组和表观基因组特征,使用机器学习方法预测了CTCF敲低后的结合持久性(图1)。研究团队收集了从3,128名患者肺组织、乳癌和前列腺癌分离的LNCaP、MCF7和IMR90细胞系的综合分子数据集,这些数据包括全基因组测序(WGS)、CTCF ChIP-seq和RNAi介导的CTCF敲低ChIP-seq数据等,将P-CTCF-BSs定义为CTCF敲低后基本不变的CTCF ChIP-seq峰位置,而L-CTCF-BSs则是CTCF ChIP-seq峰丢失的位置。
研究团队依据LNCaP和MCF7细胞系的实验数据,量化了之前研究中的特征以及新特征(n=15)在区分P-CTCF-BSs/L-CTCF-BSs方面的能力。结果显示,排名前3的特征分别是:每个ChIP-seq峰reads的富集倍数(fold enrichment)、基序评分和组成性结合。研究团队还观察到,与L-CTCF-BS相比,更多的P-CTCF-BS位于染色质环锚点、TAD边界、晚期复制时序区域,并具有更高的保守性评分。
随后,研究团队使用LNCaP数据作为训练集开发逻辑回归和随机森林模型,利用基因组和表观基因组数据预测P-CTCF-BS,并开发了一个名为CTCF-INSITE的工具。
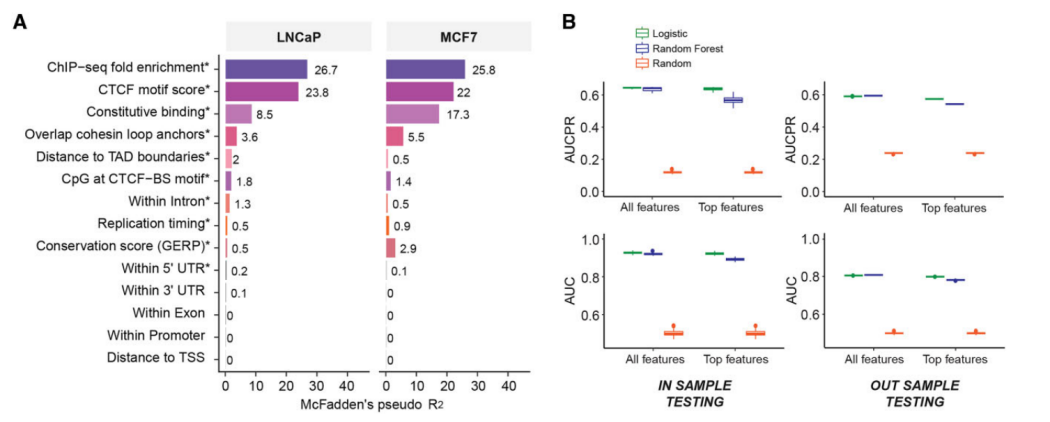
图1.开发CTCF-INSITE以预测CTCF结合持久性
P-CTCF-BSs突变率升高
研究团队通过将LNCaP和MCF7的CTCF-ChIP-seq峰区域与国际癌症基因组联盟(ICGC)WGS数据(前列腺癌和乳腺癌)中的所有突变进行交叉比对,探究了持久性和突变率之间的关系(图2)。结果显示,大部分CTCF-ChIP-seq峰区域包含≥1个突变;P-CTCF-BS比L-CTCF-BS具有更高的突变率,在LNCaP和MCF7中分别高1.34和1.25倍。特别地,P-CTCF-BS突变富集不是由位置特异性效应驱动,仅部分受到乘客突变的影响。
接下来,研究团队在LNCaP和MCF7细胞中评估了CTCF-INSITE的性能,发现其预测的P-CTCF-BS突变率与实验定义的P-CTCF-BS突变率相当,且稳健性较强。此外,染色质环锚点共定位和高CTCFChIP-seq结合强度这两个特征与高突变率相关。上述结果表明,乳腺癌和前列腺癌中的这些热点可能主要由P-CTCF-BS驱动。
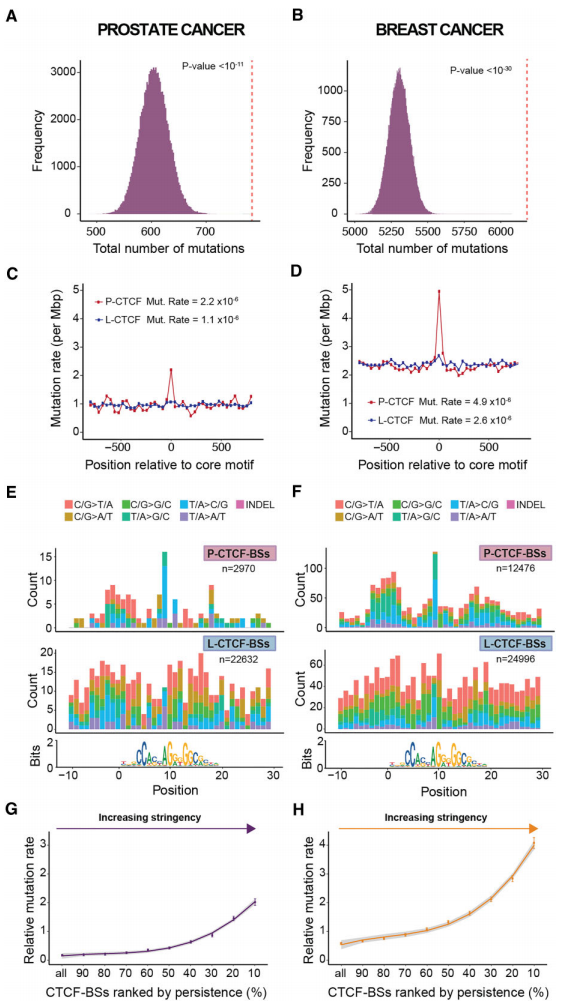
图2.P-CTCF-BS是前列腺癌和乳腺癌的突变热点
P-CTCF-BSs突变在潜在3D基因组失调位点富集
为进一步了解P-CTCF-BSs在癌症中的功能重要性,研究团队检测了P-CTCF-BSs和L-CTCF-BSs中功能性突变的富集情况,并使用ChIP-seq数据或等位基因特异性基序评分差异(Δscore)评估了CTCF结合的变化(图3)。结果显示,|Δscore|≥2的突变是定义“破坏性”突变的适当临界值,如果ICGC突变导致|Δscore|≥2,就将其归类为“破坏性”突变。在P-CTCF-BSs亚群中,共鉴定出40.4%的破坏性突变,而在L-CTCF-BSs中仅鉴定出26.5%的破坏性突变。
为证明基序评分能准确预测CTCF结合亲和力的破坏,研究团队使用重组截短CTCF蛋白(编码DNA结合结构域)进行了荧光极化DNA结合(FPDB)体外实验。结果显示,与L-CTCF-BSs相比,P-CTCF-BSs在前列腺癌和乳腺癌中都富含破坏性突变和染色质环锚定位;27%被破坏的CTCF-BS具有潜在功能,其同样位于环锚上;预测具有破坏性的候选P-CTCF-BSs突变降低了CTCF结合。值得注意的是,相较L-CTCF-BSs,P-CTCF-BSs中功能性突变的富集程度更高,表明其在癌症的3D基因组失调中起着重要作用。
研究团队汇编了乳腺癌和前列腺癌中突变P-CTCF-BS上下游1kb范围内的基因,并进行了基因集富集分析。结果显示,在乳腺癌中富集了紫外线照射(UV)后下调基因,而在前列腺癌中富集了与上皮细胞-间质转化相关的基因。这些结果表明,P-CTCF-BS突变导致CTCF结合缺失,从而导致癌症相关基因失调。
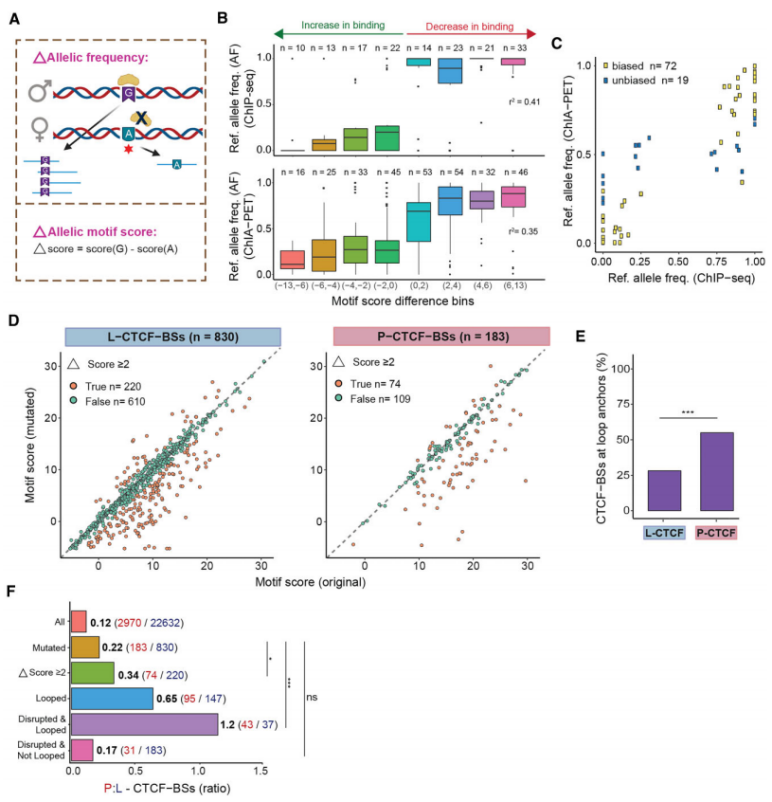
图3.等位基因分析发现P-CTCF-BS处富集功能突变
P-CTCF-BSs突变在多种癌症类型中富集
最后,研究团队评估了所观察到的P-CTCF-BSs突变富集是否是一种泛癌现象;收集了来自ICGC不同癌症队列WGS的简单突变,进行质量控制排除微卫星不稳定的癌症数据,并根据起源组织将队列中的突变分为12种癌症类型。研究团队还整理了每种癌症类型的ENCODE CTCF ChIP-seq数据,并使用CTCF-INSITE预测前10%持久性最强的CTCF-BSs,用于泛癌症富集分析。
结果表明,不同癌症类型的CTCF-BSs内的突变率有很大差异,但所有癌症的P-CTCF-BS突变率均显著高于L-CTCF-BS。有趣的是,P-CTCF-BS突变的发生率与总体突变负荷无关。此外,在大多数癌症类型中,P-CTCF-BSs也显示出破坏性突变的显著富集,这与我们在乳腺癌和前列腺癌中的观察结果类似。上述发现证实,P-CTCF-BSs是泛癌背景下的突变热点。
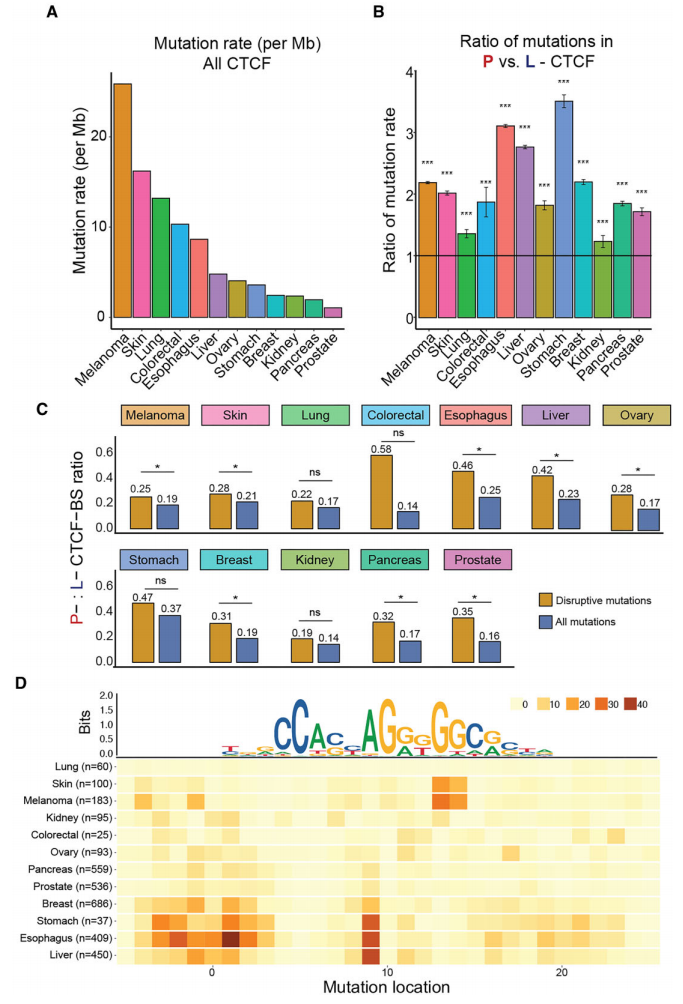
图4.P-CTCF-BS是泛癌突变热点
综上所述,研究团队开发了一种机器学习工具CTCF-INSITE,可预测P-CTCF-BSs,提供有关癌症病因学的新生物学见解;并证实P-CTCF-BSs是癌症中的突变热点,这些突变可使癌细胞具有生存优势,使其能够增殖和扩散。这一重要研究发现有潜力引领癌症诊断和治疗领域的一次革命,或将为医生们提供早期发现癌症的新手段,并为多种癌症类型带来有效的新治疗方法,显著提高癌症患者的生存质量和生存率。
参考文献:
Chen W, Zeng YC, Achinger-Kawecka J, et al. Machine learning enables pan-cancer identification of mutational hotspots at persistent CTCF binding sites. Nucleic Acids Res. Published online July 2, 2024. doi:10.1093/nar/gkae530
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言


















#癌症# #人工智能# #非编码DNA#
66