Cell:李蔚/崔亚团队首次建立串联重复扩增的全基因组图谱TR-gnomAD
2024-05-02 测序中国 测序中国 发表于上海
美国加州大学李蔚教授和崔亚教授团队合作建立了第一个biobank级别的人类多种族TR扩增全基因组图谱TR-gnomAD,包含了来自不同祖源WGS样本的0.86百万个TR。
基因组聚集数据库(gnomAD)被广泛认为是人类遗传变异的黄金标准参考图谱,但它在很大程度上忽略了串联重复序列(TR)的扩增。构成人类基因组约6%的数百万个TR对进化和人类疾病具有深远影响。与SNV和SV相比,TR具有数个数量级更高的突变率。重要的是,TR扩增与50多种致命人类疾病相关,包括肌萎缩性侧索硬化症(ALS)、亨廷顿病和多种癌症。此外,通过全基因组测序(WGS)捕获的TR扩增已被广泛用于罕见疾病的诊断。这凸显了建立TR扩增参考图谱的迫切需求,以为人类遗传变异提供全面的理解。
令人惊讶的是,迄今为止唯一的TR扩增参考图谱是从有限样本中构建的,例如来自1000基因组计划和H3Africa队列的3,350名个体。利用WGS数据的biobank规模资源(如gnomAD)在很大程度上忽视了TR扩增。这意味着,虽然TR对单基因和复杂遗传疾病的贡献巨大,但研究人员对biobank规模下TR扩增谱的理解仍然非常有限。
美国加州大学李蔚教授和崔亚教授团队合作建立了第一个biobank级别的人类多种族TR扩增全基因组图谱TR-gnomAD,包含了来自不同祖源WGS样本的0.86百万个TR。TR-gnomAD利用不同祖源之间TR单位数量频率的差异,为祖先特异性疾病的流行率提供了重要的见解。此外,TR-gnomAD还能够区分常见的、可能是良性的TR扩增(在TR-gnomAD中普遍存在)与潜在的致病性TR扩增(在疾病群体中更为频繁)。总而言之,TR-gnomAD是研究人员和医生解释遗传病患者中TR扩增的宝贵资源。该结果发表Cell上,文章题为“A genome-wide spectrum of tandem repeat expansions in 338,963 humans”。

TR-gnomAD是一个生物库规模的TR扩增参考数据库,基于338,963个人类全基因组的数据构建,平均WGS数据覆盖率为33×。这些基因组代表了不同的祖源,包括欧洲、非洲、西班牙裔、东亚和南亚等主要人群,其中39.5%(13万)为非欧洲人群数据(图1)。研究团队使用了两种准确且广泛使用的TR基因分型器,ExpansionHunter和GangSTR,以增加TR基因分型的覆盖范围。这一策略使研究团队能够对每个个体的91万个TR进行基因分型,最终保留了86万个TR进行进一步分析。分析发现,30.5%的TR至少具有两种常见等位基因(频率≥0.05)(图1D)。巨大的样本规模和多样的祖源使TR-gnomAD成为TR扩增的关键参考资源。
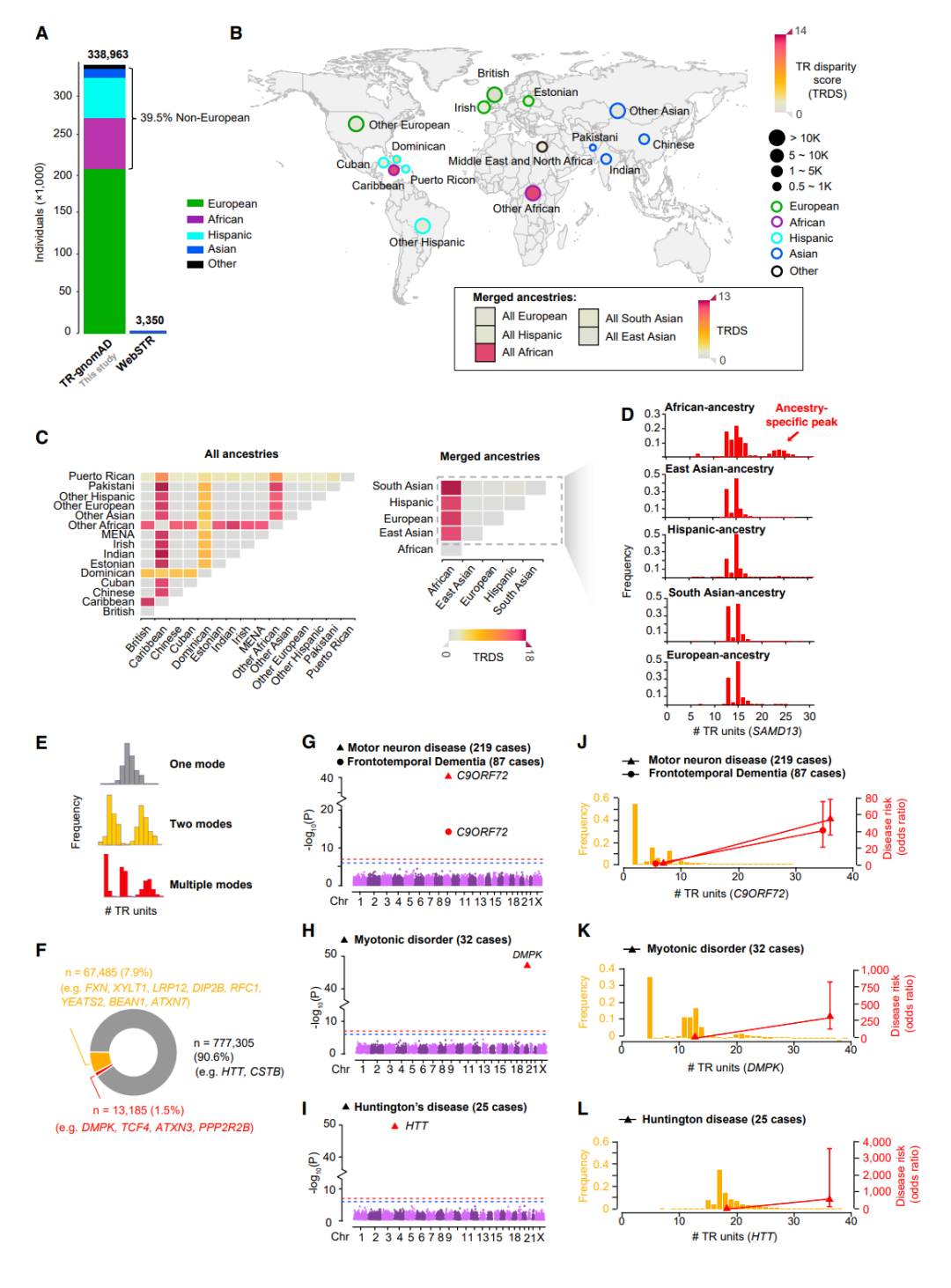
图1:来自338,963个不同祖源个体WGS基因组的TR扩增图谱。
TR-gnomAD使用户能够确定特定TR扩增在其相应祖源中的普遍性或罕见性。此外,TR-gnomAD通过在不同祖源之间TR单位数频率的差异提供了对特定祖源疾病患病率的重要洞察(图2)。研究团队开发了TR差异分数(TRDS)来量化两组人类样本TR单位数频率分布之间的差异。TR-gnomAD浏览器通过交互式页面显示了这些TRDS,比较了每个祖源与所有TR-gnomAD样本之间的差异。研究发现,至少有4.0%的TR在至少一个祖源中具有较高的TRDS(>5.0)。超过9.6%的TR在其TR单位数分布中具有多个模式(峰值),其中许多TR被发现具有致病性。特定祖源中扩增TR单位的增加频率可能直接影响与祖源相关的TR相关疾病或特征的患病率。此外,该分析揭示了相同祖源(欧洲)来自不同队列(英国生物库和TOPMed)的样本之间TRDS较小,不同祖源样本之间观察到的TRDS较大,表明将多个队列组合在一起对TRDS几乎没有影响。
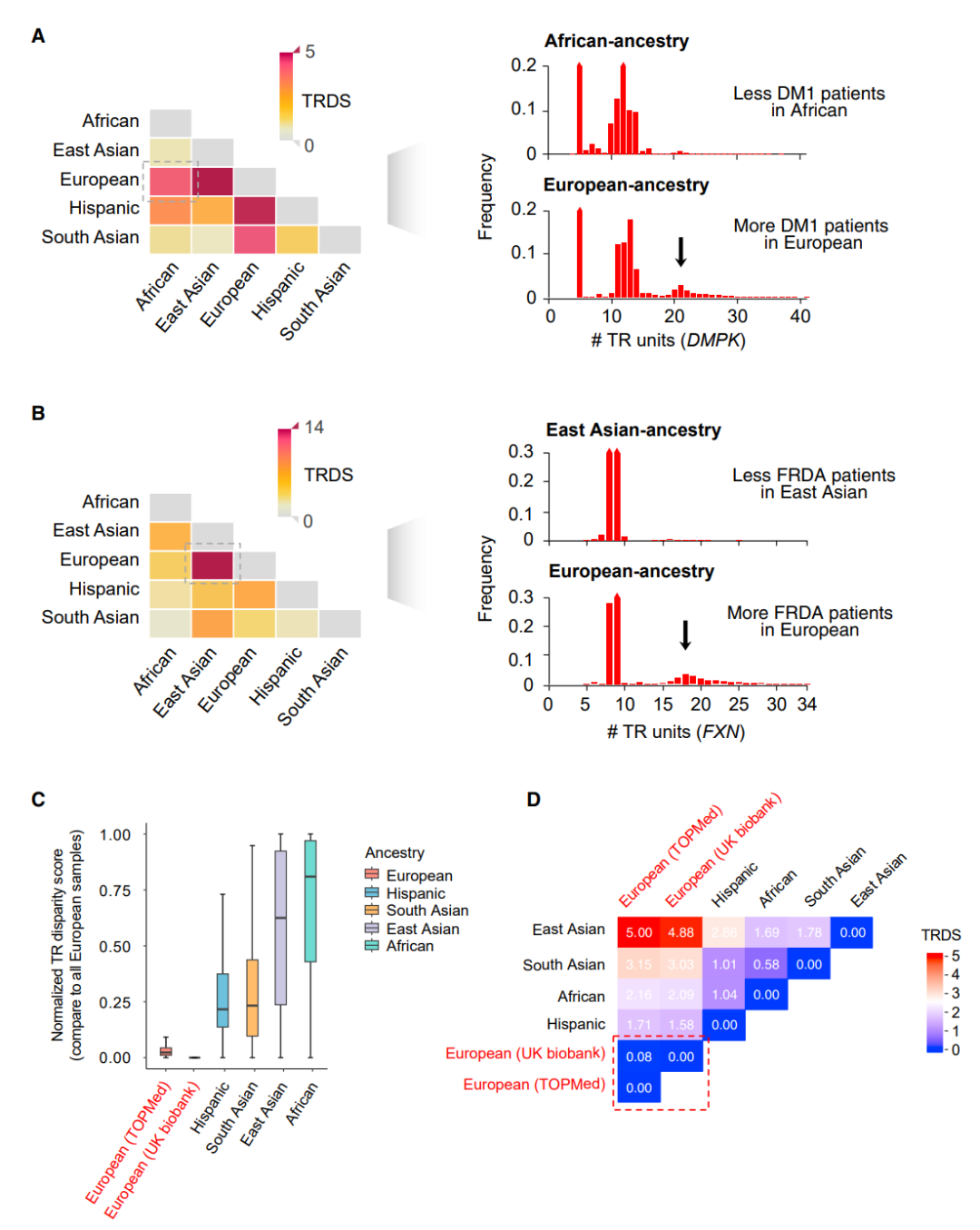
图2:不同样本之间的示例TR重复单位数量分布和TR差异分数。
最后,研究团队评估了TR-gnomAD作为解释已知临床致病性TR的对照队列的能力。利用ExpansionHunter对UK Biobank中四种TR相关罕见疾病的欧洲样本的17万个TR进行了基因分型。基于TR的全基因组关联研究和卡方检验证实了所有四种已知致病性TR与相关疾病的关联。此外,携带HTT编码TR扩增(>30个拷贝)的个体患亨廷顿病的风险较高。总的来说,以上结果强调了TR-gnomAD作为改善临床诊断工具的潜在效用。
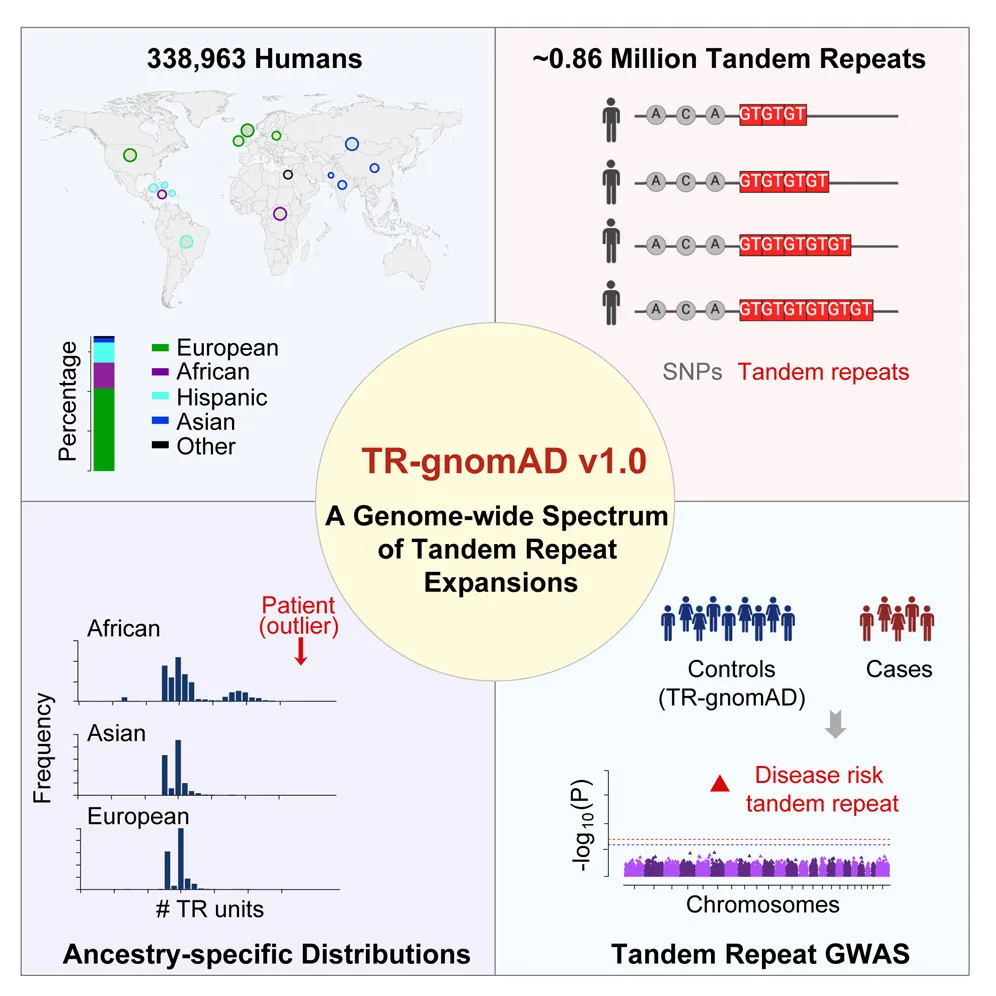
综上所述,研究团队开发的TR-gnomAD可通过不同祖源之间TR单位数频率的差异,提供对特定祖源疾病患病率的重要洞察,并区分普遍存在的、可能是良性的TR扩增与潜在致病性TR扩增。TR-gnomAD对公众免费开放,将极大的促进TR在人类遗传病中的研究分析,为遗传病患者获得遗传诊断和治疗带来新的希望。对于TR-gnomAD项目的下一阶段,研究团队计划对更多的TR进行基因分型,并优先考虑通过长读长测序确定为疾病相关的TR。
TR-gnomAD网站:
https://wlcb.oit.uci.edu/TRgnomAD
论文原文:
Ya Cui, Wenbin Ye, Jason Sheng Li, Jingyi Jessica Li, Eric Vilain, Tamer Sallam, Wei Li. A genome-wide spectrum of tandem repeat expansions in 338,963 humans. Cell, 2024. https://doi.org/10.1016/j.cell.2024.03.004
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









#TR-gnomAD# #全基因组图谱# #基因组聚集数据库#
72