academic radiology:多序列MR放射组学特征预测脑转移瘤中的EGFR T790M突变
2023-11-07 shaosai MedSci原创 发表于上海
放射组学是一个不断发展的领域,通过从图像中提取一组特征,可以将医学图像自动分类到一个预定义的群体中。
据统计,肺癌(LC)仍然是全世界癌症相关发病率和死亡率的主要原因。表皮生长因子受体(EGFR)是非小细胞肺癌(NSCLC)中最常见的致癌驱动突变。现阶段,第一代或第二代表皮生长因子受体酪氨酸激酶抑制剂(TKIs)已被确定为治疗携带表皮生长因子受体突变的NSCLC患者最有希望的药物之一。然而,几乎所有最初对这些TKIs有显著反应的患者最终都会通过各种机制产生获得性耐药。体细胞突变,即苏氨酸790与蛋氨酸(T790M)的替换,大约占一半是耐药的主要原因。除了获得性T790M突变外,原发性T790M突变偶尔会在NSCLC患者中检测到,这些患者对第一代或第二代TKIs也不敏感。中枢神经系统(CNS)是LC最常见的转移部位,也是治疗后复发的初始部位,与病情进展不佳有关。一些研究结果表明,在接受第一代或第二代TKIs治疗的EGFR突变患者中,T790M与脑转移(BM)明显相关。奥希美替尼是第三代TKIs,可以同时针对EGFR激活突变以及T790M耐药突变,包括原发性和获得性T790M。因此,在NSCLC病程中评估EGFR突变情况,早期识别T790M耐药性突变,特别是对于疾病进展的患者来说可以及时调整靶向治疗策略、意义重大。
现阶段,临床上确认基因型的金标准仍然是通过活检进行病理检查。但大多数T790M突变是在EGFR-TKIs治疗期间由肿瘤细胞获得的,其检测需要对基因事件进行分析。鉴于活检的侵入性,反复获取样本是不可行的。此外,穿刺活检通常只对病变的一小部分进行量化,可能会受到肿瘤异质性的影响。循环肿瘤DNA(ctDNA)可作为评估T790M突变的替代方法,但受限于循环血液中的低丰度和高成本。
磁共振成像(MRI)是诊断和评估BM的基石,然而分子和遗传信息是肉眼看不见的。因此,开发一个非侵入性的、可重复的影像学标志物,实现整个肿瘤病变的评估以预测T790M耐药性突变,将具有很大的意义。放射组学是一个不断发展的领域,通过从图像中提取一组特征,可以将医学图像自动分类到一个预定义的群体中。一些证据表明,基于转移性肿瘤的放射组学可预测肿瘤的基因型。然而据我们所知,还没有研究评估过基于BM的多序列MRI放射组学在识别T790M耐药突变方面的价值。
近日,发表在academic radiology杂志的一项研究开发并验证了一个用于识别有BM的NSCLC的EGFR T790M突变的基于多序列MRI的机器学习模型,并进一步探讨了哪种磁共振序列能达到最佳的诊断性能,为临床的进一步准确的进行风险评估。
本项回顾性研究招募了233名NSCLC患者,其中包括来自两家医院的95名T790M患者和138名非T790M患者,分别作为训练队列和测试队列。分别从T2WI、T2流体增强反转恢复(T2-FLAIR)、扩散加权成像(DWI)和对比增强T1加权成像(T1-CE)序列中提取放射组学特征。根据最大信息系数和Boruta方法,选择了最可预测的特征。然后通过随机森林分类器建立四个放射组学模型来描述T790M突变的特征。构建ROC曲线、F1得分和DCA曲线,以验证四个模型的能力和性能。
DWI模型表现出最好的性能,在训练队列中AUC和F1分数为0.886和0.789,在测试队列中为0.850和0.743。DCA曲线也显示,在测试队列中,DWI模型的整体净效益高于其余三个模型。其他三个模型无论在训练或测试队列中也有一定的分类能力,特别是T2-FLAIR模型。
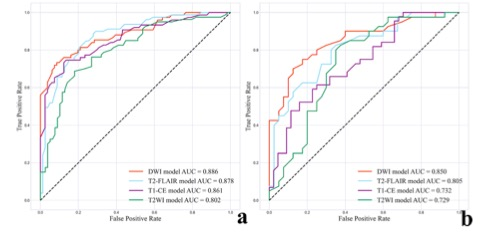
图 DWI、T2-FLAIR、T1-CE和T2WI模型的ROC曲线。(a) 训练队列,(b) 测试队列
本项研究表明,基于多序列MRI的放射组学可预测EGFR T790M耐药突变的出现,特别是基于DWI序列的放射组学特征价值更高。
原文出处:
Ye Li,Xinna Lv,Bing Wang,et al.Predicting EGFR T790M Mutation in Brain Metastases Using Multisequence MRI-Based Radiomics Signature.DOI:10.1016/j.acra.2022.12.030
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言