全基因组甲基化研究技术原理介绍
2014-02-05 MedSci MedSci原创
DNA甲基化是基因组DNA的一种主要表观遗传修饰形式。近年来,大量研究表明DNA甲基化修饰对于维持正常细胞功能、传递基因组遗传印记、胚胎发 育以及人类肿瘤发生,起着至关重要的作用。甲基化研究成为表观遗传学的热点,相应的技术也是层出不穷,根据实验目的的不同,这些技术大体能够分成两类:全 基因组(高通量)甲基化研究技术以及特异性甲基化位点(低通量)研究技术。下面将介绍几种当前最常用的一些技术全
DNA甲基化是基因组DNA的一种主要表观遗传修饰形式。近年来,大量研究表明DNA甲基化修饰对于维持正常细胞功能、传递基因组遗传印记、胚胎发
育以及人类肿瘤发生,起着至关重要的作用。甲基化研究成为表观遗传学的热点,相应的技术也是层出不穷,根据实验目的的不同,这些技术大体能够分成两类:全基因组(高通量)甲基化研究技术以及特异性甲基化位点(低通量)研究技术。下面将介绍几种当前最常用的一些技术全基因组或者说中高通量甲基化研究手段,供大家参考:
芯片平台:
基于芯片平台的全基因组甲基化分析无疑是性价比最高的甲基化图谱分析方式,目前市场上主流的实验平台主要是illumina和agilent,相应的产品
包括illumina人的商品化芯片(HumanMethylation27 DNA analysis BeadChip,
HumanMethylation450 DNA analysis BeadChip),agilent人和小鼠的商品化芯片(Human CpG
Island Microarray,Human DNA Methylation Microarray以及Mouse CpG Island
Microarray),过去Roche NimbleGen也有相应的产品,但从今年下半年已经停产。
使用不同的实验平台,操作方法也不同,illumia芯片前期处理采用的是亚硫酸盐处理,将甲基化的差异转变成碱基上的差异,然后利用两个位点特异的探针
检测这些化学上差异的位点,一个探针是为甲基化位点(M磁珠类型)设计的,而另一个是为未甲基化位点(U磁珠类型)设计的,结合后通过单碱基延伸掺入一个
标记了的ddNTP,通过计算甲基化位点和未甲基化位点掺入的荧光信号强度检测DNA甲基化程度。该项技术被称为Infinium技术,即单碱基延伸技术,包括InfiniumI和InfiniumII,示意图如下:
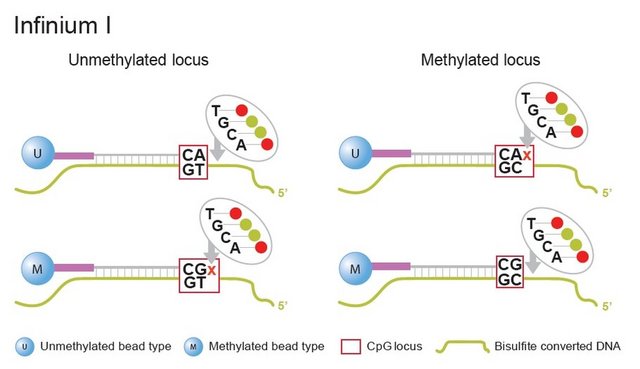
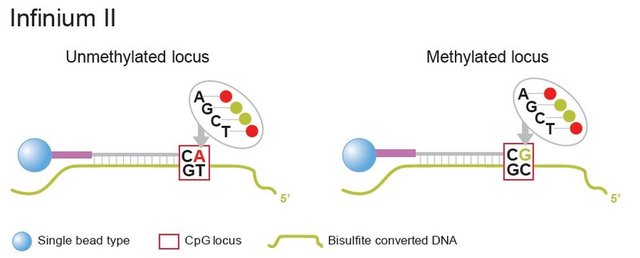
illunina最经典的产品莫过于HumanMethylation27 DNA analysis
BeadChip,该芯片含有>27,578个CpG位点,产品性能和评价都很高。不过,随着研究逐步的深入,这款产品已经不能满足研究的需要,慢
慢被另一款产品HumanMethylation450 DNA analysis
BeadChip取代,包含>450,000个CpG位点,该产品最大的特点在于覆盖范围广,而且除了CpG岛,还涵盖有CpG
shores和CpG
shlves区域,以及non-CpG位点以及miRNA启动子区域等,无疑是DNA全基因组甲基化研究的又一利器,在对基因组non-CpG位点研究
[1]以及在Avon Longitudinal Study of Children and Parents (ALSPAC)
[2]中都用到这款芯片。而goleden
gate是illumina的定制芯片平台,适合于样本量大,中高通量的甲基化研究。芯片以矩阵微珠芯片(Sentrix Array
Matrix(SAM))的格式,每个panel可以检测96个样本,根据客户的定制要求,每个样品可同时检测384-1536个甲基化位点。
而安捷伦采用的是免疫共沉淀的方法(MeDIP-chip)的方法。基因组DNA分成两份,一份用来做MeDIP,加入抗5'-甲基化胞嘧啶核苷抗体,使
用免疫磁珠法分离样品中甲基化DNA片段的抗体复合物,样品中其余的非甲基化DNA片段被洗脱;另一份作为对照。两个样品都标记荧光(富集的样品
input用Cy5标记,对照用Cy3标记),然后与芯片杂交。芯片上每个探针的Cy5/Cy3强度比例显示出该区域的甲基化程度。
agilent Human CpG Island
Microarray规格为1x244K,包含约195000个探针检测27800个CpG岛。Human DNA Methylation
Microarray规格为1x244K,能够检测27627个扩展了的CpG岛(expanded CpG
islands)和5081个甲基化不做的区域(UMR region), Mouse CpG Island
Microarray规格为2x105K,能够检测16030个CpG位点。agilent
还有著名的earray平台,能够根据客户要求对芯片进行定制[3]。
二代测序平台:
新一代测序仪的飞速发展,使得测序成本大幅度下降,也使得甲基化组(methylome)的研究成为可能。近两年,多个研究小组利用高通量测序相结合,绘制出了多张甲基化图谱。
提到基于二代测序平台的全基因组研究,首先想到的必然是“金标准”BS-seq,即全基因组重亚硫酸盐测序(whole genome
bisulfite
sequencing)。该方法的实验原理是前期用Bisulfite处理,将基因组中未发生甲基化的C碱基转换成U,进行PCR扩增后变成T,与原本具
有甲基化修饰的C碱基区分开来,再结合高通量测序技术,特别适用于绘制单碱基分辨率的全基因组DNA甲基化图谱。但是相应的费用也是相当高的。为了能用较
低的成本得到全面的基因组甲基化状况,有两种性价比非常高的测序方法不得不提,那就是MeDIP-seq(甲基化DNA免疫共沉淀测序)和RRBS(简化
的表观亚硫酸氢盐测序技术(Reduced Representation Bisulfite Sequencing))。
MeDIP-seq是基于抗体富集原理进行测序的全基因组甲基化检测技术,采用甲基化DNA免疫共沉淀技术,通过5'-甲基胞嘧啶抗体特异性富集基因组上
发生甲基化的DNA片段,然后通过高通量测序可以在全基因组水平上进行高精度的CpG密集的高甲基化区域研究。该方法优点在于精度高,覆盖广,性价比高,
但是不足之处就是不能精确到单碱基分辨率,这能知道一段区域的甲基化程度。
而RRBS则是基于酶切原理。2005年Alexander Meissner等在Nucleic Acids
Research首次提出该方法[4],之后被许多研究人员采用[1,5]。该方法前期用MspI酶切,该酶的酶切位点为CCGG,从而将CpG位点富集出来,最终用较小的数据量得到包含最多CpG位点甲基化信息的单碱基精度的甲基化图谱。相应的技术路线如下图所示。
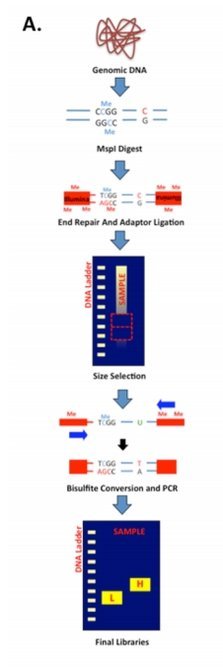
[1] Ziller MJ, Muller F, Liao J, et al. Genomic Distribution and Inter-Sample Variation of Non-CpG Methylation across Human Cell Types. PLoS Genet, 2011, 7(12): 1-15.
[2] Abigail Fraser,Corrie Macdonald-Wallis,Kate Tilling, et al. Cohort Profile: The Avon Longitudinal Study of Parents and Children: ALSPAC mothers cohort International Journal of Epidemiology 2012,1-14.
[3]LIAN Zhen-Qiang ; QI WANG ; LI Wen-Ping, et al. Screening of significantly hypermethylated genes in breast cancer using microarray-based methylated-CpG island recovery assay and identification of their expression levels. Int J Oncology. 2012,627-636.
[4]Alexander Meissner, Andreas Gnirke, George W. Bell, et al. Reduced representation bisulfite sequencingfor comparative high-resolution DNAmethylation analysis. Nucleic Acids Research. 2005,33(18):5868-5877.
[5] Zachary D. Smith, Hongcang Gu, Christoph Bock, et al. High-throughput bisulfite sequencing in mammalian genomes.Methods,2009,48(3): 226-232.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言









#全基因组#
50